El sitio etiquetado de secuencia, y el mapeo STS, es otra técnica de mapeo físico.
#mapeodegenes, #STS, #NCBI, #ADN, #biomedicina
Un STS es una secuencia corta de ADN que se ha demostrado que es única Para calificar como un STS, la ubicación exacta el orden de las bases de la secuencia deben ser conocidos esta secuencia puede ocurrir solo una vez en el cromosoma que se está estudiando, y en el genoma en su totalidad si el conjunto de fragmentos de ADN cubre todo el genoma.
#mapeodegenes, #STS, #NCBI, #ADN, #biomedicina
Un STS es una secuencia corta de ADN que se ha demostrado que es única Para calificar como un STS, la ubicación exacta el orden de las bases de la secuencia deben ser conocidos esta secuencia puede ocurrir solo una vez en el cromosoma que se está estudiando, y en el genoma en su totalidad si el conjunto de fragmentos de ADN cubre todo el genoma.
Fuentes comunes de STSs
Las etiquetas de secuencia expresada (EST) son secuencias cortas obtenidas por análisis de clones de ADN complementario (ADNc). El ADN complementario se prepara convirtiendo el ARNm en ADN de doble cadena se cree que representa las secuencias de los genes que se expresan. Una EST se puede usar como STS si proviene de un gen único no de un miembro de una familia de genes en la que todos los genes tienen las mismas secuencias similares.
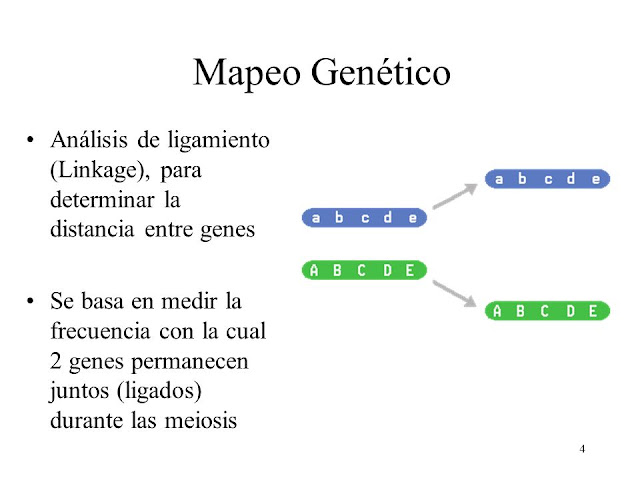
Los polimorfismos de longitud de secuencia simple (SSLP) son matrices de secuencias repetidas que muestran variaciones de longitud Los SSLP que son polimórficos que ya han sido mapeados por el análisis de vinculación son particularmente valiosos ya que proporcionan una conexión entre los mapas genéticos físicos.
Las secuencias genómicas aleatorias se obtienen mediante la secuenciación de fragmentos aleatorios de ADN genómico clonado mediante el examen de secuencias ya depositadas en una base de datos.
Para mapear un conjunto de STS, una colección de fragmentos de ADN superpuestos de un cromosoma se digiere en fragmentos más pequeños utilizando enzimas de restricción, agentes que cortan las moléculas de ADN en puntos objetivo definidos. Los datos partir de los cuales se derivará el mapa se obtienen al observar qué fragmentos contienen qué STS. Para lograr esto, los científicos copian los fragmentos de ADN mediante un proceso conocido como clonación molecular ". La clonación implica el uso de una tecnología especial, llamada tecnología de ADN recombinante, para copiar fragmentos de ADN dentro de un huésped extraño. Primero, los fragmentos se unen con un portador, y un vector. Luego de la introducción en un huésped adecuado, los fragmentos de ADN se pueden reproducir junto con el ADN de la célula huésped, proporcionando material ilimitado para el estudio experimental. Un conjunto no ordenado de fragmentos de ADN clonados se denomina biblioteca.
A continuación, los clones, y copias, se ensamblan en el orden en que se encontrarían en el cromosoma original al determinar qué clones contienen fragmentos de ADN superpuestos. Este conjunto de clones superpuestos se denomina cóntigo de clon. Una vez que se conoce el orden de los clones en un cromosoma, los clones se colocan en un almacenamiento congelado la información sobre el orden de los clones se almacena en una computadora, lo que proporciona un recurso valioso que se puede usar para estudios posteriores. Luego, estos datos se utilizan como material base para generar una secuencia de ADN larga continua, y los STS sirven para anclar la secuencia en un mapa físico.
La necesidad de integrar mapas físicos genéticos
Al igual que con la mayoría de las técnicas complejas, el mapeo basado en STS tiene sus limitaciones. Además de las brechas en la cobertura de los clones, los fragmentos de ADN pueden perderse o asignarse erróneamente a una posición incorrecta. Estos errores pueden ocurrir por una variedad de razones. Un fragmento de ADN puede romperse, lo que resulta en un STS que se asigna una posición diferente. Los fragmentos de ADN también pueden eliminarse de un clon durante el proceso de replicación, lo que resulta en la ausencia de un STS que debería estar presente. A veces, un clon compuesto de fragmentos de ADN de dos regiones genómicas distintas se replica, lo que lleva que los segmentos de ADN que están ampliamente separados en el genoma se asignen erróneamente a posiciones adyacentes. Por último, un fragmento de ADN puede contaminarse con material genético del huésped, lo que nuevamente lleva a un STS que se asignará a la ubicación incorrecta. Para ayudar superar estos problemas, así como para mejorar la precisión general del mapeo, los investigadores han comenzado a comparar e integrar mapas físicos basados en STS con mapas genéticos, de radiación híbrida citogenéticos. Las referencias cruzadas de diferentes mapas genómicos mejoran la utilidad de un mapa dado, confirman el orden de STS y ayudan a ordenar y orientar los cóntigos en evolución.
NCBI la integración de mapas
La comparación de los muchos mapas genéticos físicos disponibles puede llevar mucho tiempo, especialmente cuando se trata de identificar la ubicación de un nuevo gen. Sin el uso de computadoras software especial diseñado para alinear los distintos mapas, sería muy difícil hacer coincidir una secuencia con una región de un cromosoma que corresponde a la ubicación del gen. Sería como tratar de comparar 20 mapas interestatales callejeros diferentes para llegar desde una casa en Ukiah, California, y una casa en Beaver Dam, Wisconsin. Puede comparar los mapas usted mismo y crear su propio itinerario de viaje, pero probablemente tomará mucho tiempo ¿No sería más fácil y rápido que el club del automóvil cree un mapa integrado para usted? Ese es el objetivo detrás del Visor de mapas del genoma humano del NCBI.
NCBI: Centro Nacional para la Información Biotecnológica o National Center for Biotechnology Information. Forma parte de la Biblioteca Nacional de Medicina de los Estados Unidos (NLM), una rama de los Institutos Nacionales de la Salud (NIH). El NCBI está ubicado en Bethesda, Maryland y se fundó en 1988 a través de una legislación patrocinada por el Senador Claude Pepper.
Visor de mapas del NCBI: una herramienta para integrar mapas genéticos físicos
NCBI presentó recientemente a la comunidad científica una nueva herramienta de software llamada Entrez Map Viewer. El Visor de mapas proporciona una visualización gráfica de los datos disponibles de la secuencia del genoma humano, así como los mapas híbridos citogenéticos, genéticos, físicos de radiación. El Visor de mapas puede mostrar simultáneamente hasta siete mapas, seleccionados de un conjunto de 19, y permite al usuario acceder información detallada para una región de mapa seleccionada. El Visor de mapas utiliza un sistema de numeración de secuencias común para alinear los mapas de secuencia los marcadores compartidos, así como los nombres de los genes para alinear otros mapas. Puede usar el Visor de mapas de NCBI para buscar un gen en varios genomas, y se están agregando más genomas con regularidad.
Referencias: https://camstanbookspain.blogspot.com/p/enfermedades-geneticas-clasificacion.html

Comentarios
Publicar un comentario