Los marcadores de ADN se pueden utilizar para investigación básica (por ejemplo, análisis filogenético y el estudio de genes útiles) e investigación aplicada (por ejemplo, selección mediante marcadores, identificación de paternidad y seguimiento de alimentos).
#RFLP, #marcadoresADN, #VNTR, #SNP
Los RFLP o polimorfismos de longitud de fragmentos de restricción, fueron algunos de los primeros marcadores de ADN desarrollados. Los RFLP se definen por la presencia ausencia de un sitio específico, llamado sitio de restricción, para una enzima de restricción bacteriana. Esta enzima rompe hebras de ADN siempre que contengan una cierta secuencia de nucleótidos.
#RFLP, #marcadoresADN, #VNTR, #SNP
Los RFLP o polimorfismos de longitud de fragmentos de restricción, fueron algunos de los primeros marcadores de ADN desarrollados. Los RFLP se definen por la presencia ausencia de un sitio específico, llamado sitio de restricción, para una enzima de restricción bacteriana. Esta enzima rompe hebras de ADN siempre que contengan una cierta secuencia de nucleótidos.
Los VNTR o número variable de polimorfismos de repetición en tándem, se producen en regiones no codificantes del ADN. Este tipo de marcador se define por la presencia de una secuencia de nucleótidos que se repite varias veces. En cada caso, el número de veces que se repite una secuencia puede variar.
Los polimorfismos de microsatélite se definen por un número variable de repeticiones de un número muy pequeño de pares de bases dentro de una secuencia. A menudo, estas repeticiones consisten en los nucleótidos, y bases, citosina adenosina. El número de repeticiones para un microsatélite dado puede diferir entre individuos, de ahí el término polimorfismo: la existencia de diferentes formas dentro de una población.
Los SNP, o polimorfismos de un solo nucleótido, son mutaciones puntuales individuales, y sustituciones de un solo nucleótido, que no cambian la longitud total de la secuencia de ADN en esa región. Los SNP se producen en todo el genoma de un individuo.
Del análisis de ligamiento al mapeo genético
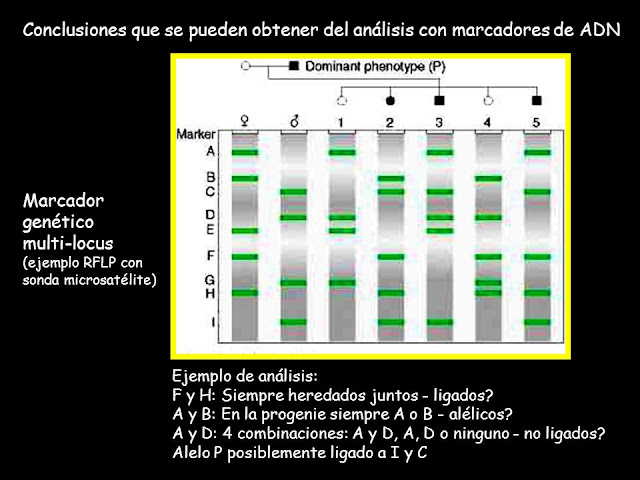
Los primeros genetistas reconocieron que los genes están ubicados en los cromosomas creían que cada cromosoma individual se heredaba como una unidad intacta. Ellos plantearon la hipótesis de que si dos genes estaban ubicados en el mismo cromosoma, se unían físicamente se heredaban juntos. Ahora sabemos que este no es siempre el caso. Los estudios realizados alrededor de 1910 demostraron que muy pocos pares de genes mostraban un vínculo completo. Los pares de genes se heredaron de forma independiente mostraron un vínculo parcial, es decir, se heredaron juntos veces, pero no siempre.
Durante la meiosis, el proceso mediante el cual se producen los gametos (óvulos, espermatozoides), dos copias de cada par de cromosomas se acercan físicamente. Los brazos del cromosoma pueden sufrir una ruptura intercambio de segmentos de ADN, un proceso denominado recombinación cruce Si ocurre una recombinación, cada cromosoma encontrado en el gameto consistirá en una "mezcla" de material de ambos miembros del par de cromosomas. Por lo tanto, los eventos de recombinación afectan directamente el patrón de herencia de los genes involucrados.
Como uno no puede ver físicamente los eventos de cruce, es difícil determinar con cierto grado de certeza cuántos cruces han ocurrido realmente. Pero, utilizando el fenómeno de co-segregación de alelos de marcadores cercanos, los investigadores pueden revertir la meiosis identificar los marcadores que están cerca uno del otro. Luego, utilizando una técnica estadística llamada análisis de ligamiento genético, los investigadores pueden inferir un patrón de cruce probable, y de ahí un orden de los marcadores involucrados. Los investigadores también pueden inferir una estimación de la probabilidad de que se produzca una recombinación entre cada par de marcadores.
Si la recombinación ocurre como un evento aleatorio, entonces los dos marcadores que están muy juntos deben separarse con menos frecuencia que los dos marcadores que están más alejados entre sí. La probabilidad de recombinación entre dos marcadores, que puede variar de a 0,5, aumenta monótonamente a medida que aumenta la distancia entre los dos marcadores lo largo de un cromosoma. Por lo tanto, la probabilidad de recombinación se puede usar como un sustituto para ordenar marcadores genéticos lo largo de un cromosoma. Si luego determina las frecuencias de recombinación para diferentes pares de marcadores, puede construir un mapa de sus posiciones relativas en el cromosoma.
Pero, por desgracia, predecir la recombinación no es tan simple. Aunque los cruces son aleatorios, no están distribuidos uniformemente en el genoma ni en ningún cromosoma. Algunas regiones cromosómicas, llamadas puntos calientes de recombinación, tienen más probabilidades de participar en cruces que otras regiones de un cromosoma. Esto significa que la distancia del mapa genético no siempre indica la distancia física entre los marcadores a pesar de estas calificaciones, el análisis de vinculación generalmente deduce correctamente el orden de los marcadores, y las estimaciones de distancia son suficientes para generar mapas genéticos que pueden servir como un marco valioso para la secuenciación del genoma.
Comentarios
Publicar un comentario