La secuenciación SMRT se basa en que la secuenciación por síntesis esté a mano. El DNA se sintetiza en guías de onda de modo cero (ZMW), pequeños contenedores en forma de pozo con las herramientas de captura ubicadas en el fondo del pozo. La secuenciación se realiza con el uso de polimerasa sin modificar (unida al fondo de ZMW) y nucleótidos marcados con fluorescencia que fluyen libremente en la solución. Los pocillos están construidos de manera que solo se detecte la fluorescencia que se produce en el fondo del pozo. La etiqueta fluorescente se desprende del nucleótido tras su incorporación en la cadena DNA, dejando un DNA sin modificar DNA .hebra. Según Pacific Biosciences (PacBio), el desarrollador de la tecnología SMRT, esta metodología permite la detección de modificaciones de nucleótidos (como la metilación de citosina). Esto sucede a través de la observación de la cinética de la polimerasa. Este estar a la mano permite análisis de 20 000 nucleótidos o más, con longitudes de lectura promedio de 5 kilobases. En 2015, Pacific Biosciences anunció el lanzamiento de un nuevo instrumento de secuenciación llamado Sequel System, con 1 millón de ZMW en comparación con 150, 000 ZMW en el instrumento PacBio RS II. La secuenciación SMRT se conoce como secuenciación de "tercera generación" o "lectura larga".
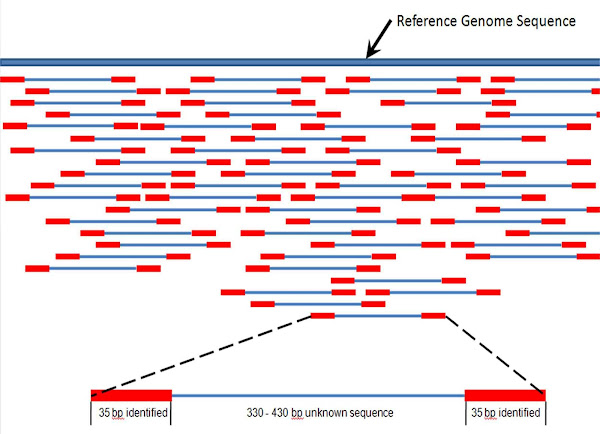
Imagen 151A | Los análisis de concatenación múltiples y fragmentados deben ensamblarse sobre la base de sus áreas superpuestas. | Suspencewl / CC0 | Page URL : (https://commons.wikimedia.org/wiki/File:Mapping_Reads.png) de Wikimedia Commons
Autor : Milos Pawlowski
Comentarios
Publicar un comentario