Se desarrollaron varios métodos nuevos para la secuenciación de DNA a mediados y finales de la década de 1990 y se implementaron en secuenciadores comerciales de DNA en el año 2000. En conjunto, estos métodos se denominaron secuenciación de "próxima generación" o "segunda generación"( NGS) métodos, en la regulación para divulgarlos de los métodos anteriores antes mencionados, como Sanger Secuenciación. Mientras que para la primera generación de secuenciación, NGS La tecnología suele caracterizarse por ser altamente escalable, lo que permite secuenciar todo el genoma a la vez. Habitualmente, esto se logra fragmentando el genoma en trozos pequeños, muestreando aleatoriamente un fragmento y secuenciando utilizando una de una variedad de tecnologías, como se muestra en las que se describen a continuación. Un genoma completo es posible sobre la base de que se secuencian múltiples fragmentos a la vez (lo que le da la denominación de secuenciación "masivamente paralela) en una operación automatizada.
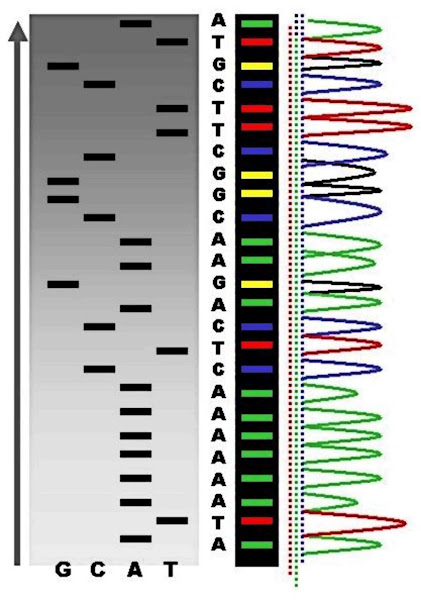
Imagen 149A | Un ejemplo de los resultados de la secuenciación automática de terminación de cadena DNA. | Abizar en Wikipedia en inglés / CC BY-SA (http://creativecommons.org/licenses/by-sa/3.0/legalcode) | Page URL : (https://commons.wikimedia.org/wiki/File:Radioactive_Fluorescent_Seq.jpg) de Wikimedia Commons
Autor : Milos Pawlowski
Comentarios
Publicar un comentario