El uso de plásmidos como técnica en biología molecular está respaldado por software bioinformático. Estos programas registran la concatenación DNA de los vectores plasmídicos, ayudan a predecir los sitios de corte de las enzimas de limitación y a planificar las manipulaciones. Ejemplos de paquetes de software que manejan mapas de plásmidos son ApE, Clone Manager, GeneConstructionKit, Geneious, Genome Compiler, LabGenius, Lasergene, MacVector, pDraw32, Serial Cloner, VectorFriends, Vector NTI y WebDSV. Estas piezas de software ayudan a realizar experimentos completos in silico antes de realizar experimentos húmedos.
Colecciones de plásmidos
Se han creado muchos plásmidos a lo largo de los años y los investigadores han entregado plásmidos a bases de datos de plásmidos, por ejemplo, las organizaciones sin fines de lucro Addgene y BCCM / LMBP. Se pueden identificar y solicitar plásmidos de esas bases de datos para la investigación. Además, los investigadores a menudo cargan secuencias de plásmidos en la base de datos NCBI, desde donde se pueden recuperar secuencias de plásmidos específicos.
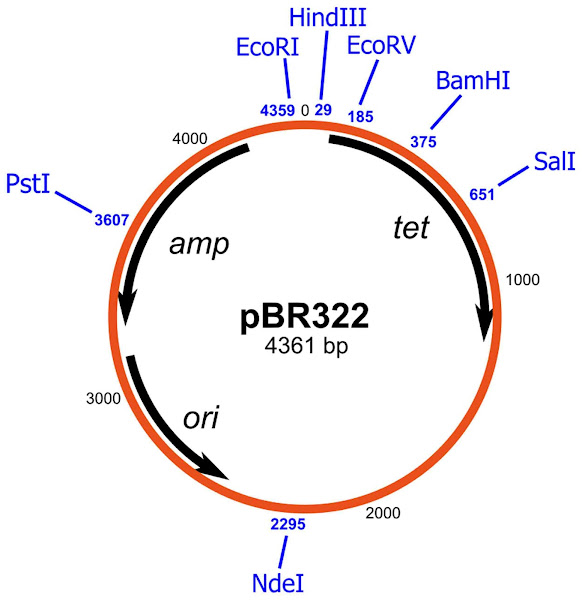
Imagen 239A | Una representación esquemática del vector pBR322 con los sitios de limitación indicados en azul. | Ayacop (+ Yikrazuul) / Public domain | Page URL : (https://commons.wikimedia.org/wiki/File:PBR322.svg) from Wikimedia Commons
Autor : Milos Pawlowski
Comentarios
Publicar un comentario