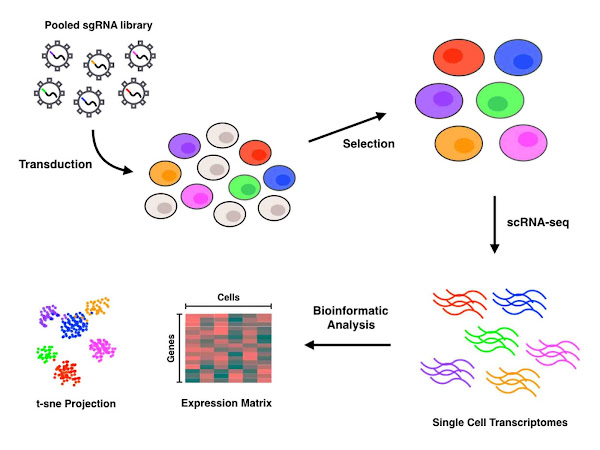
Una vez que se han seleccionado células transducidas con éxito, es necesario aislar células individuales para realizar scRNA-seq. Perturb-seq y CROP-seq se han realizado utilizando tecnología basada en gotas para el aislamiento de una sola célula, por otro lado, el CRISP-seq estrechamente relacionado se realizó con una extracción basada en micropocillos. Una vez que las células se han aislado a nivel de una sola célula, tiene lugar la copia inversa, la amplificación y la secuenciación para producir la forma genética de los perfiles de expresión para cada célula. Muchos enfoques de scRNA-seq incorporan identificadores moleculares únicos (UMI) y códigos de barras de celda durante el paso de copia inversa para indexar al individuo RNA moléculas y células, respectivamente. Estos códigos de barras adicionales sirven para ayudar a cuantificar las transcripciones de RNA y para asociar cada una de las secuencias con su célula de origen.
Imagen 240A | Descripción general del flujo de trabajo Perturb-seq | Sdlee94 / Attribution-Share Alike 4.0 International | Page URL : (https://commons.wikimedia.org/wiki/File:Overview_of_Perturb-seq_workflow.jpeg) from Wikimedia Commons
Autor : Milos Pawlowski
Comentarios
Publicar un comentario