En la secuenciación de microfluidos Sanger toda la amplificación por termociclado de los fragmentos DNA, además, ya que su separación por electrophoresis se realiza en una sola oblea de vidrio (aproximadamente 10 cm de diámetro) reduciendo así el uso de reagent como coste. En algunos casos, los investigadores han demostrado que pueden aumentar el rendimiento de la secuenciación convencional mediante el uso de microchips. Aún será necesario realizar investigaciones en materia de reglamentación para que este uso de la tecnología sea eficaz.
Técnicas basadas en microscopía
Esta a mano visualiza directamente la concatenación de moléculas DNA mediante microscopía electrónica. La primera identificación de pares de bases DNA dentro de moléculas DNA intactas mediante la incorporación enzimática de bases modificadas, que contienen átomos de número atómico aumentado, visualización directa e identificación de bases marcadas indicativamente dentro de una molécula sintética de 3, 272 pares de bases DNA y se ha demostrado un genoma viral de 7, 249 pares de bases.
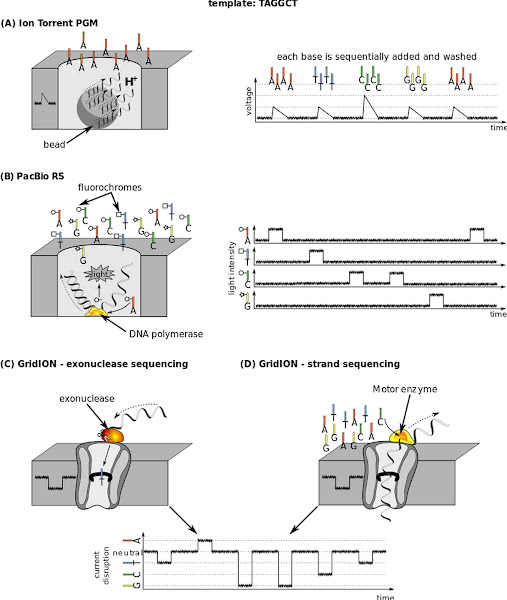
Imagen 153A | Secuenciación de la plantilla TAGGCT con IonTorrent, PacBioRS y GridION | Philippe Hupé / CC BY-SA (https://creativecommons.org/licenses/by-sa/3.0/legalcode) | Page URL : (https://commons.wikimedia.org/wiki/File:From_second_to_fourth-generation_sequencing,_illustration_on_TAGGCT_template.svg) de Wikimedia Commons
Autor : Milos Pawlowski
Comentarios
Publicar un comentario